Utilities
» NMR関連
-
Co-ANAFOR (201218版)
- Co-ANAFORは、多次元NMR解析において、限られたポイント数のデータから、実測していないデータを再構成して、高分解能のスペクトルを得る方法である。
- Linear Prediction等の従来法と比べると、別の高分解能スペクトルから得られた化学シフトの情報を利用する点が特徴であり、蛋白質の2Dスペクトルのような、シグナルが混んでいるデータでも、正確なシグナル強度比を算出することが可能である。
- 詳細な原理や他の手法との比較に関しては、参考文献を参照。
必要なプログラム
- Bruker Topspin
- Anaconda (pythonの数値解析ライブラリ(numpy, scipy等)をまとめてインストール・管理するプラットフォーム)
- NMRスペクトル解析用ソフト(Sparky等)
Co-ANAFORスクリプトのダウンロード
- Python3.X用
- 2Dスペクトル再構成用のスクリプト:201218python3_COANAFOR2D.py
- 3Dスペクトル再構成用のスクリプト:201220python3_COANAFOR3D.py
- Python2.X用
- 2Dスペクトル再構成用のスクリプト:201217python2_COANAFOR2D.py
- 3Dスペクトル再構成用のスクリプト:201219python2_COANAFOR3D.py
マニュアルのダウンロード
- ここをクリックすると、日本語のマニュアルのダウンロードが始まります。
-
SARS-COV2 NMRデータベース
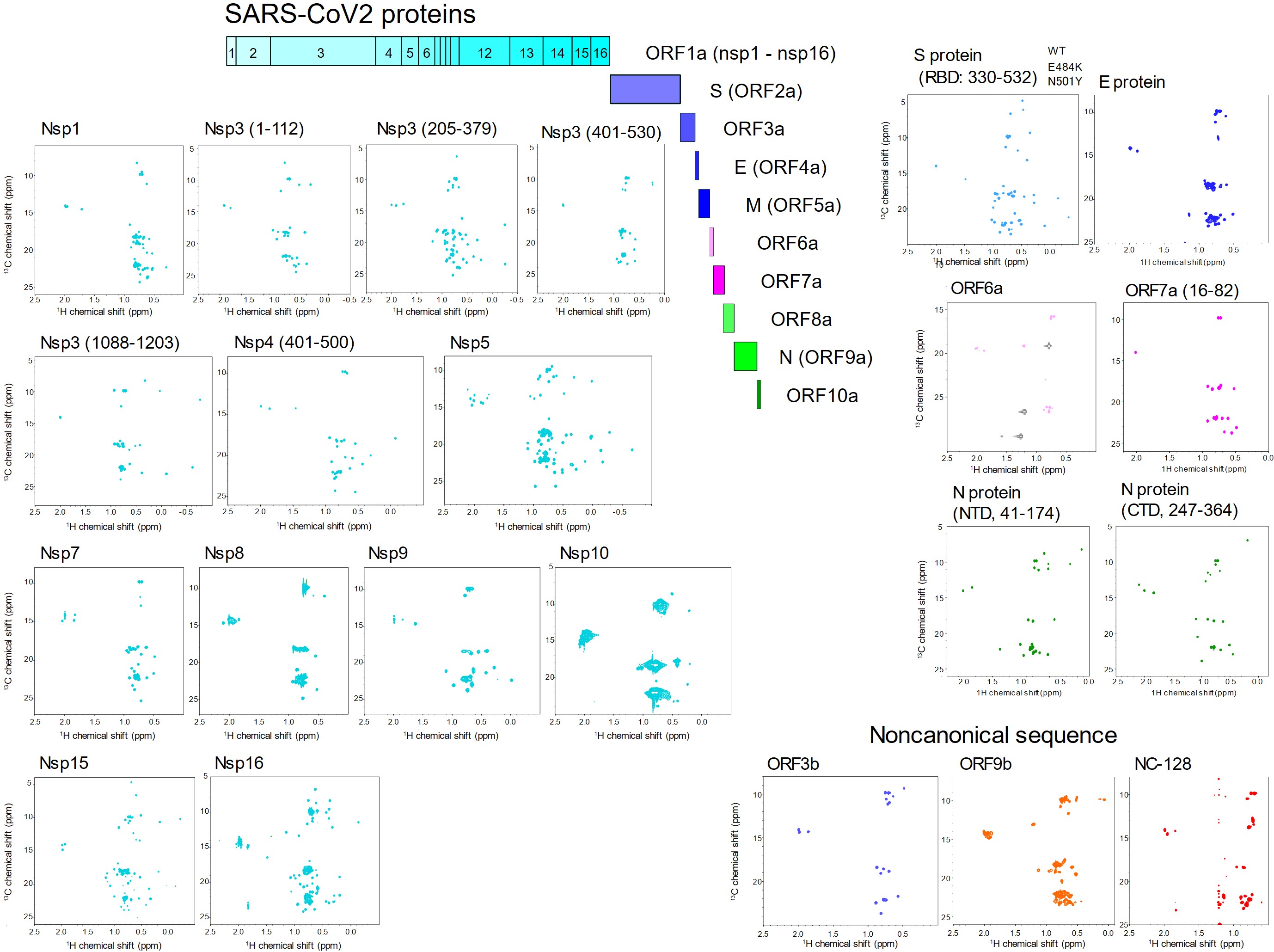
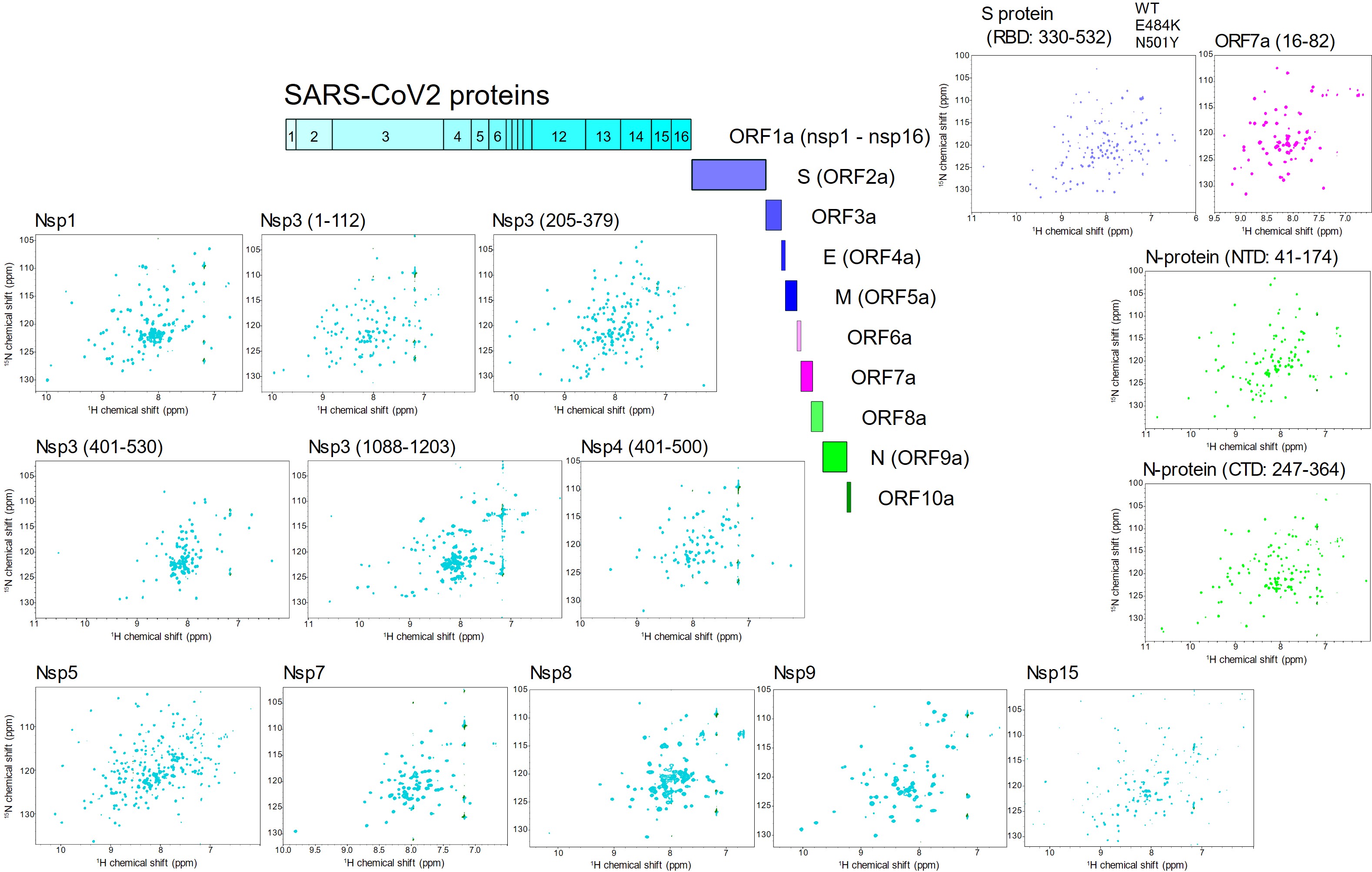
産業技術総合研究所(産総研)細胞分子工学研究部門は、AMEDプロジェクト「新型コロナウイルス感染症等対処技術開発プロジェクト」の一環として、SARS-CoV-2タンパク質の45の構造ドメインのうち、27ドメインの無細胞発現系による発現と精製に成功した。これらはウイルスに含まれるORFの50%以上をカバーしている。また、ORF3bやORF9bのような非定型タンパク質の取得にも成功した。
SARS-COV2-NMRデータベースは、22個の構造ドメイン(Table I)の2次元NMRスペクトルから構成されており、3つの非定型タンパク質(Table II、Ref.1)を含んでいる。これらは、精製タンパク質の構造について均一性と品質を保証するデータとなっている。このスペクトルにより、SARS-CoV-2タンパク質の相互作用や構造を原子レベルで解析することが可能となり、創薬や生物学的基礎研究に役立つことが期待される。
(2025年より東京大学に移管)13C Ile, Leu, Met, Val methyl TROSY spectra (クリックすると各スペクトルを確認できます※PCのみ)

S protein WT E484K N501Y15N TROSY spectra (クリックすると各スペクトルを確認できます※PCのみ)

S protein WT E484K N501Y
-
» その他
-
ギャラリー
準備中
-